[本站讯]近日,山东大学软件学院吴昊副教授团队在长非编码RNA亚细胞定位研究领域取得新进展,相关成果以“Lnclocator-imb: An Imbalance-tolerant Ensemble Deep Learning Framework for Predicting Long Non-coding RNA Subcellular Localization”为题发表在生物医学健康领域国际顶尖学术期刊IEEE Journal of Biomedical and Health Informatics(中科院1区, Top期刊, IF = 7.7分)。软件学院硕士研究生刘海斌为本文的第一作者,吴昊副教授为该论文唯一通讯作者,山东大学软件学院为第一作者单位和唯一通讯作者单位。该篇学术论文的发表是学校近年来促进多学科交叉建设方面取得高质量科研成果的一个缩影。
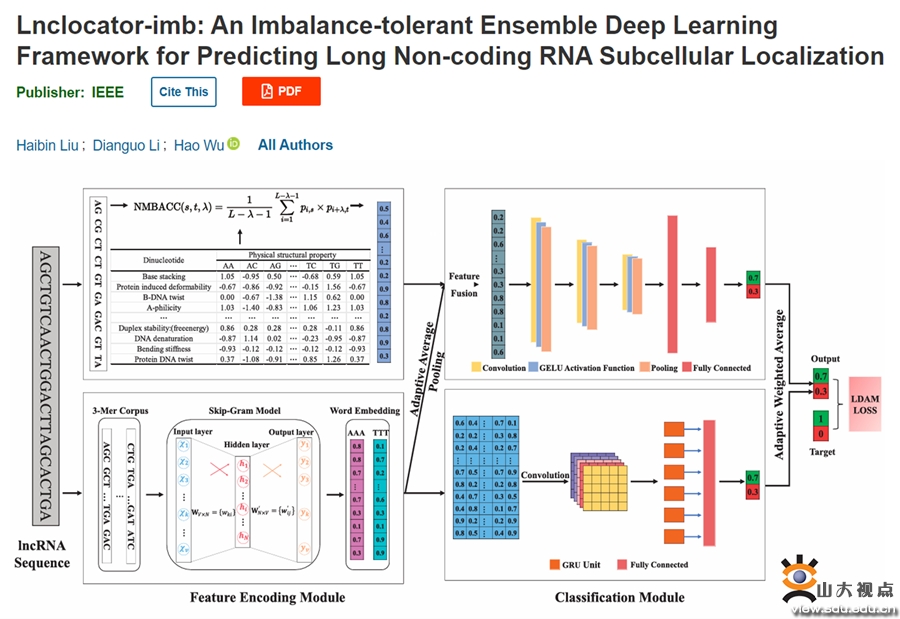
长非编码RNA(lncRNA)在各种生物过程中起着关键作用,包括剂量补偿、表观遗传调控、细胞周期调控和细胞分化调控等,因此,lncRNA已成为遗传研究领域的核心问题。确定lncRNA的亚细胞定位对于获得lncRNA相互作用、后转录和共转录调控修饰以及直接影响lncRNA功能的外部因素至关重要。当前,随着数据量的急剧增多,基于计算的方法已成为预测lncRNA亚细胞定位的有效途径。然而,在处理不平衡数据集时,当前计算方法的性能还需要进一步提升。
本研究结合物理化学模式特征和核酸的分布表示特征提出了一种集成深度学习模型lncLocator-imb,用于预测lncRNA亚细胞定位。为有效解决模型在面对不平衡数据集时性能不佳的问题,我们在训练过程中使用了标签分布感知边界(LDAM)损失函数。此外,本研究全面评估了lncLocator-imb和先前研究方法的性能,全面验证了lncLocator-imb的有效性和优越性能。此外,本研究使用SHAP(SHapley Additive exPlanations,沙普利加和解释)进行特征贡献和依赖性分析,分析结果为特征选择的合理性提供了证据,并确定了可能对lncRNA亚细胞定位产生潜在影响的二核苷酸组,为生物学家提供借鉴和参考。此外,本研究提出了一种管理不同特征和解决不平衡数据集的新框架,该框架有望成为各种基于序列预测任务的重要资源,为生物信息学和遗传学领域的专业人士提供了一个多功能预测工具。
吴昊副教授团队长期致力于复杂疾病通路、三维基因组结构、调控元件预测、亚细胞定位和单细胞多组学数据集成及下游分析等相关研究,近期的系列研究成果发表于Nucleic Acids Research(2022,中科院1区, IF=19.16),Briefings in Bioinformatics(2022a, 2022b, 2022c,中科院1区, IF=13.99),Bioinformatics(2022,中科院1区, IF=6.93),IEEE Journal of Biomedical and Health Informatics(2023a, 2023b,中科院1区, IF=7.7),Computers in Biology and Medicine(2023,中科院1区, IF=7.7)等学术期刊,相关研究得到国家自然科学基金,国家重点研发计划等项目资助。
论文链接:https://ieeexplore.ieee.org/document/10286044。
